|
Asma
INTRODUÇÃO
Clonagem Posicional
Existem duas formas para a identificação de genes.
● Uma inicia-se com uma proteína conhecida, determina-se
a sequência de aminoácidos e se empregam estas informações para isolar o
gene.
● A outra identifica genes por sua posição no genoma (Clonagem
Posicional).
A clonagem posicional é uma estratégia utilizada para identificar
genes e sua posição relativa no genoma e o seu produto protéico. A clonagem
posicional não necessita de nenhum conhecimento prévio da função do gene. A
clonagem posicional baseia-se apenas na localização cromossômica do gene.
Essencialmente, envolve o "screening" de todo o DNA, analisado
cuidadosamente através de milhares de fragmentos de DNA. São necessárias duas
instâncias para mapear genes de uma doença:
● Um número suficiente de famílias, com a doença bem documentada, para estabelecer a ligação (linkage) e, ligação (linkage) e,
● Marcadores genéticos adequados de DNA.
A segunda exigência pode ser encontrada com relativa facilidade
na atualidade pois, o projeto humano do genoma identificou milhares de marcadores
úteis do DNA. Uma vez identificadas as famílias, os investigadores determinam
se as pessoas doentes na família apresentam sequências particulares de DNA
em posições específicas, não encontradas nos seus membros saudáveis. Este
processo é feito através da coleta de sangue (10 ml) dos membros da família,
da análise genética dos marcadores de DNA, através de técnicas que utilizam
a PCR (polymerase chain reaction) e análises de sequenciamentos automatizadas
de genes entre os membros doentes e saudáveis da família, através de máquinas
computadorizadas com potente capacidade em "bioinformática".
Assim um mapeamento genético de ligação revelará quais genes
são os principais suspeitos e a sequência dos mesmos que poderá ser então
analisada diretamente em pacientes individuais.
Um marcador particular do DNA é correlacionado à doença (linked)
se, em geral, membros da família com certos nucleotídeos no marcador sempre
apresentam a doença e membros da família com outros nucleotídeos no marcador
não apresentam a doença. 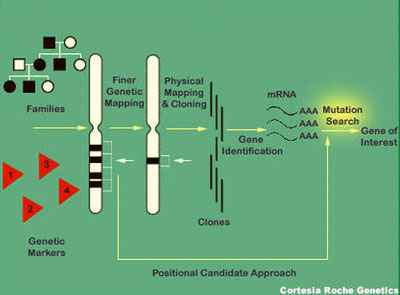
Quando surgem evidências para uma determinada região
de um cromossomo, esta região deve ser decomposta para aproximadamente 1 Mb
(megabase) (1 Mb = 1 000 000 pares de base). Para isto, a região requer saturação
com um grande número de marcadores  polimórficos,
previamente estudados em indivíduos com asma. Após decompor a região, torna-se
possível procurar genes que possam estar implicados na doença, por clonagem
posicional. Isola-se o gene e efetua-se o teste para a presença de mutações
particulares que conferem características da doença, sendo a região DNA clonada
através de uma variedade de vetores de clonagem, como por exemplo, o polimórficos,
previamente estudados em indivíduos com asma. Após decompor a região, torna-se
possível procurar genes que possam estar implicados na doença, por clonagem
posicional. Isola-se o gene e efetua-se o teste para a presença de mutações
particulares que conferem características da doença, sendo a região DNA clonada
através de uma variedade de vetores de clonagem, como por exemplo, o  YAC, abreviatura em inglês para yeast
artificial chromosomes (cromossomomo artificial de levedura). YAC, abreviatura em inglês para yeast
artificial chromosomes (cromossomomo artificial de levedura).
Os vetores que contêm fragmentos do DNA da região de interesse
são isolados verificando-os para ver se há a presença dos marcadores polimórficos
para lócus de ligação. Estes fragmentos contribuem para um mapa genético
da região chamada de  contig de YAC. A ordem dos clones é definida por pequenas sequências de DNA,
chamadas de STS (sequence-tagged site). Tendo-se construído
um contig, os genes podem ser isolados por uma variedade de métodos: p. ex:
o YACs que mapeia a região de interesse pode ser rotulado com radioatividade
e usado diretamente como sonda para a análise das bibliotecas de DNA
complementar (cDNA). contig de YAC. A ordem dos clones é definida por pequenas sequências de DNA,
chamadas de STS (sequence-tagged site). Tendo-se construído
um contig, os genes podem ser isolados por uma variedade de métodos: p. ex:
o YACs que mapeia a região de interesse pode ser rotulado com radioatividade
e usado diretamente como sonda para a análise das bibliotecas de DNA
complementar (cDNA).
|